
Science Advances | 白凡/王俊团队合作揭示亚实性肺腺癌结节肿瘤微环境特征
随着低剂量电子计算机断层扫描(Low-dose Computed Tomography, LDCT) 技术在肺癌筛查中的普及,肺结节的检出比例大幅度提高[1]。依据在CT下能否完全遮盖肺实质可将肺结节分为实性和亚实性两类,而亚实性结节(Subsolid Nodule, SSN)又可根据有无实性成分进一步分为磨玻璃结节和部分实性结节两类。SSN是一种影像学上的非特异表现,恶性SSN病理特征可能是浸润前或者浸润性肺腺癌(Lung Adenocarcinoma, LUAD)。
目前的临床共识认为影像学表现为SSN的肺腺癌是一种相对惰性的特殊亚型,具有非常好的预后[2]。肿瘤微环境(Tumor Microenvironment,TME)是一个包含多种细胞类型的复杂“生态系统”,TME的组成特点对肿瘤生物学特性的塑造至关重要,因此全面详细地刻画SSN的TME有助于理解其特点,也可能为研究肺腺癌发生发展和浸润转移提供新的思路。

针对此科学问题,北京大学人民医院胸外科主任王俊院士团队与北京大学生物医学前沿创新中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、生命科学学院白凡研究员团队开展合作研究,应用单细胞转录组测序技术全面揭示了影像学表现为SSN的肺腺癌肿瘤微环境特征,为SSN相对惰性的生物学特性和较好的临床预后特点提供了单细胞尺度上的解释。2021年1月27日,研究论文 "Decoding the multicellular ecosystem of lung adenocarcinoma manifested as pulmonary subsolid nodules by single-cell RNA sequencing" 以Research Article的形式在线发表于国际期刊Science Advances。
研究团队对16名CT影像学表现为SSN的肺腺癌患者的手术切除样本进行单细胞转录组测序,同时整合了已经发表的6例癌旁样本(nLung)和9例进展性肺腺癌样本(mLUAD)的数据,通过典型相关分析(CCA)算法无偏的整合了nLung、SSN和mLUAD样本,一共获得了118,293个高质量的单细胞数据(图. 1A)。
降维聚类鉴定出10种主要的细胞类型,包括EPCAM+ 上皮细胞、T淋巴细胞、B淋巴细胞、浆细胞、髓系细胞、自然杀伤细胞(NK)、肥大细胞、成纤维细胞、内皮细胞和血红细胞(图. 1B-1C)。
通过分析CD45+的免疫细胞相对比例发现:在SSN中,肥大细胞显著富集;T细胞比例介于nLung与mLUAD之间;髓系细胞相比nLung显著减少,相反的B细胞和浆细胞在SSN中显著富集,这与mLUAD类似;NK细胞在SSN中的比例接近nLung,明显多于mLUAD (图. 1D-1E)。
以上结果说明,相比nLung与mLUAD,SSN的TME在细胞组成上已经呈现出不同的模式。
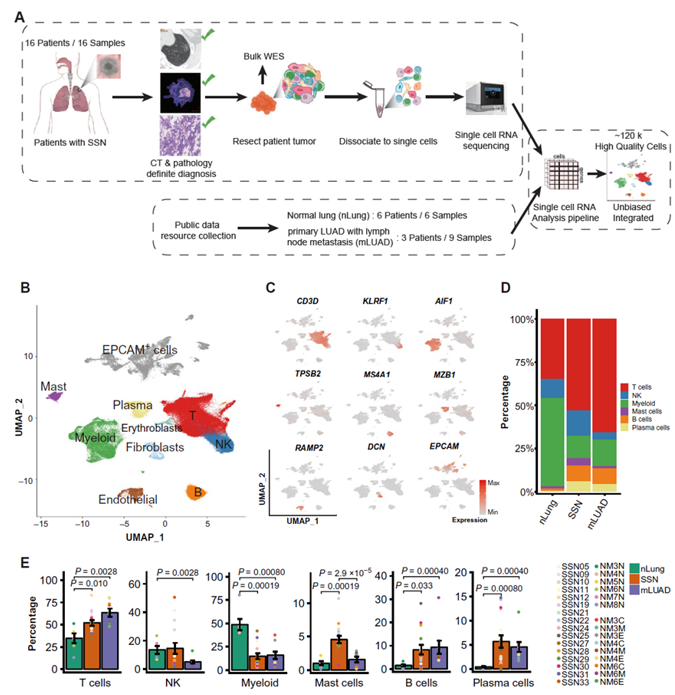
图1:研究流程示意图与SSN肿瘤微环境全景图
通过单细胞转录组推断拷贝数变异识别出的肿瘤细胞呈现出明显的肿瘤样本间异质性(图. 2A-2B)。相比于SSN,mLUAD肿瘤细胞明显上调细胞增殖、干扰素响应、PI3K-AKT和乏氧等相关的通路。相比于正常上皮,SSN肿瘤细胞除了上调细胞增殖相关的通路外,还明显上调很多代谢相关的通路,包括葡萄糖代谢、脂肪酸代谢和氧化磷酸化等(图. 2C)。通过计算代谢通路活性发现,SSN肿瘤细胞呈现独特的代谢模式(图. 2D)。肿瘤样本内异质性分析发现mLUAD中存在细胞周期、线粒体信号、上皮分化、上皮间质转化和乏氧的功能模块。在SSN中,研究人员识别出一个强烈的免疫应激功能模块(图. 2E)。以上结果表明,相比于mLUAD,SSN中的肿瘤细胞恶性程度相对较低,呈现出明显的代谢紊乱和强烈的免疫反应应激状态。

图2:肿瘤细胞的识别与状态比较
接着,研究人员分析了SSN肿瘤免疫微环境中的各种细胞。在T/NK细胞方面,鉴定出5种CD4+ T细胞,5种CD8+ T细胞和2种NK细胞亚型(图. 3A)。SSN中效应CD4+ T,耗竭CD4+ T和调节性CD4+ T细胞比例均处在nLung和mLUAD中间(图. 3B)。SSN的CD8+ T细胞整体组成更接近nLung,与mLUAD差别很大,表现为富集杀伤性很强的效应CD8+ T,缺乏两群与LUAD患者生存显著相关的耗竭CD8+ T细胞(图. 3B-3D)。通过计算“杀伤耗竭比”,发现SSN的杀伤耗竭比值介于nLung和mLUAD中间(图. 3E)。有趣的是,研究发现相比于mLUAD,SSN中显著地富集CD16+ NK细胞亚群(图. 3B)。以上结果均说明SSN中T/NK细胞免疫功能整体处于免疫监视杀伤而非耗竭状态。
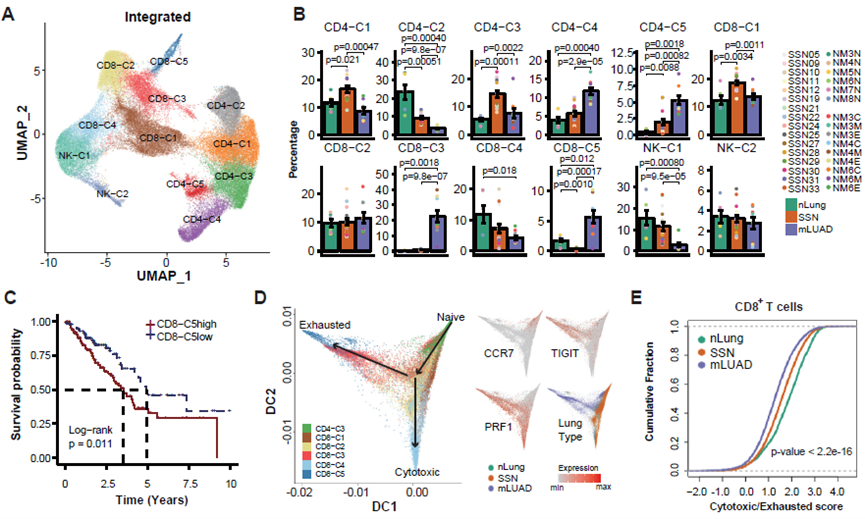
图3:T/NK细胞亚群比例变化与状态比较
髓系细胞亚群在三种不同状态下组成差异比较大,反映了髓系细胞在不同TME的复杂性和多样性(图. 4A-4B)。SSN中显著富集不同的DCs亚类,这些DCs表达不同的共刺激分子,并且SSN中DCs低表达促进CD4+ T细胞向免疫抑制性调节T细胞分化的重要基因IDO1(图. 4C)。
特别地,研究发现了一群只在mLUAD中富集并高表达VEGFA、CCL2、SLC2A3、ADM等乏氧诱导基因的肿瘤相关巨噬细胞,Macro-C7 (图. 4D),生存分析显示这群细胞与LUAD患者的预后显著相关(图. 4E)。标志基因比较发现Macro-C7与结直肠癌中报道的一群促进血管生成和EMT的巨噬细胞高度相似[3],提示其在LUAD的浸润转移中可能的作用。
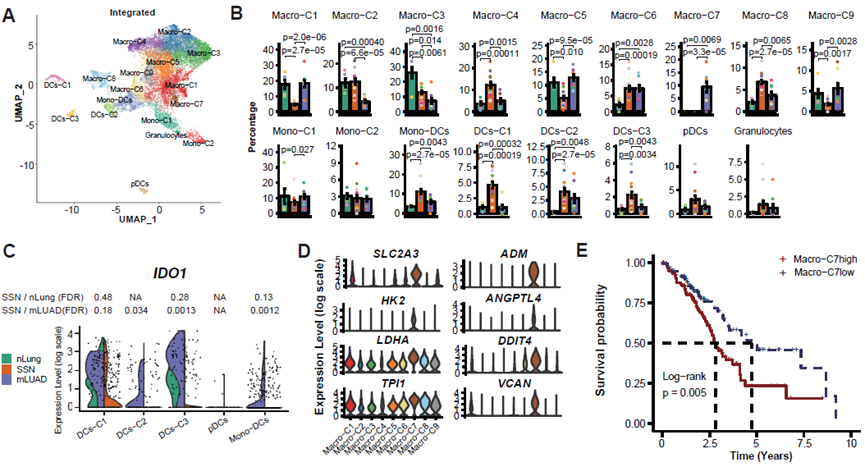
图4:髓系细胞亚群的鉴定与特征刻画
研究也对成纤维细胞和内皮细胞两种基质细胞进行了详细的刻画。一共识别出6群内皮细胞和5群成纤维细胞(图. 5A-5D)。有趣的是,SSN两种基质细胞的组成呈现两种完全相反的情况。内皮细胞的组成比例已经与mLUAD十分相似,表现为血管生成相关的Endo-C3增多,淋巴细胞归巢相关的Endo-C5的减少(图. 5B)。而SSN成纤维细胞的组成比例仍然与nLung基本一致而与mLUAD完全不同,缺少肿瘤相关成纤维Fibro-C3和周细胞Fibro-C5(图. 5D)。特别地,研究人员发现SSN中富集一群与免疫相关的成纤维细胞Fibro-C4,提示SSN中成纤维细胞可能存在免疫调节作用。以上结果说明,内皮细胞可能在LUAD早期阶段就扮演非常重要的作用。

图5:TME中基质细胞的识别与比较
最后,研究人员通过“受体-配体”分析推测细胞类型之间的相互作用。相比于nLung,SSN和mLUAD中内皮细胞和巨噬细胞的“输出事件”明显增多,提示这两种细胞在癌症进展中的促进作用。比较发现,SSN中NK细胞的整体互作事件明显多于nLung和mLUAD,这与NK细胞在SSN中富集结果一致,提示NK细胞在SSN免疫微环境中的重要作用。进一步数据分析和实验验证结果显示SSN中富集的NK细胞可能是内皮细胞招募的。
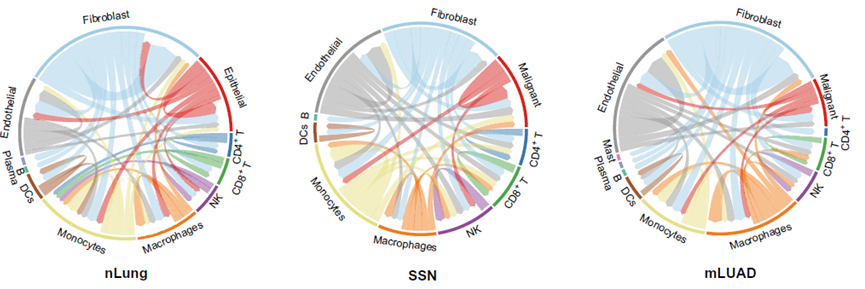
图6:细胞-细胞间相互作用的特点
该研究基于高通量单细胞转录组测序数据,详细刻画了SSN的TME中肿瘤细胞、免疫细胞、基质细胞的细胞类型,组成特点,转录组状态和细胞间互作等特征。特别的是,研究人员通过将SSN与nLung比较,揭示了SSN恶性发生发展的特点。通过与mLUAD比较,在单细胞尺度上解释了为何SSN具有相对惰性的生物学特征和较好的临床预后特点。对SSN和肺腺癌相关的分子基础研究,临床治疗和药物研发等方面具有重要意义和价值。
北京大学人民医院胸外科主任王俊院士、邱满堂助理研究员和北京大学生物医学前沿创新中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、生命科学学院白凡研究员为本文的共同通讯作者。清华大学生命科学学院、北京大学-清华大学-北京生命科学研究所联合项目博士研究生邢旭东和北京大学人民医院胸外科杨帆教授为本文的共同第一作者。
原文链接:https://advances.sciencemag.org/content/7/5/eabd9738
参考文献
[1]L. Fan, Y. Wang, Y. Zhou, Q. Li, W. Yang, S. Wang,F. Shan, X. Zhang, J. Shi, W. Chen, S. Y. Liu, Lung cancer screening withlow-dose CT: Baseline screening results in shanghai. Acad. Radiol. 26,1283–1291 (2019).
[2]F. Fu, Y. Zhang, Z. Wen, D. Zheng, Z. Gao, H. Han,L. Deng, S. Wang, Q. Liu, Y. Li, L. Shen, X. Shen, Y. Zhao, Z. Zhao, T. Ye, J.Xiang, Y. Zhang, Y. Sun, H. Hu, H. Chen, Distinct prognostic factors inpatients with stage I non-small cell lung cancer with radiologic part-solid orsolid lesions. J. Thorac. Oncol. 14, 2133–2142 (2019).
[3]L. Zhang, Z. Li, K. M. Skrzypczynska, Q. Fang, W.Zhang, S. A. O'Brien, Y. He, L. Wang, Q. Zhang, A. Kim, R. Gao, J. Orf, T.Wang, D. Sawant, J. Kang, D. Bhatt, D. Lu, C. M. Li,A. S. Rapaport, K. Perez, Y. Ye, S. Wang, X. Hu, X.Ren, W. Ouyang, Z. Shen, J. G. Egen, Z. Zhang, X. Yu, Single-cell analysesinform mechanisms of myeloid-targeted therapies in colon cancer. Cell 181,442–459.e29 (2020).