
PNAS | 一种新的基于Tn5转座酶的转录组测序快速建库方法
1月27日,北京大学的黄岩谊课题组和谢晓亮课题组联合清华大学王建斌课题组完成的一篇名为“RNA sequencing by direct tagmentation of RNA/DNA hybrids”的研究论文以直接投稿(Direct Submission)方式发表在美国《国家科学院院刊》(PNAS ) 。该文章开发了一种新的基于Tn5转座酶的转录组测序快速建库方法,与已有的其它方法相比,大大简化了建库的过程 ;同时对样品的用量还很小,不仅可以用于高质量的单细胞转录组测序,而且对包括目前正在肆意蔓延的冠状病毒等样本的测序质量和速度或许也将有大大的提升。与谢晓亮教授此前发表的MALBAC、LIANTI等方法一样,研究团队继续以红酒的名字来命名,并起名为SHERRY(Sequencing HEteRo RNA-DNA-hYbrid )。

RNA测序目前已广泛应用于绘制物种转录组图谱、研究疾病分子调控机制等方向,微量及单细胞RNA测序技术的开发更是在时间和空间维度上为RNA信息的获取提供了更高的分辨率。然而,针对不同研究需求,不同起始量的RNA需要采用不同的RNA扩增方法,且目前所公知的扩增方法多操作繁复、耗时长,不利于多样本的实验。
Tn5转座酶复合体因能一步打断双链DNA并且使DNA两端带上接头,而广泛应用于DNA测序建库技术。该论文研究发现,Tn5转座酶同样也能以类似的方式作用于RNA/DNA杂交链,从而能够省去传统RNA建库技术中必不可少的RNA打断、cDNA二链合成及扩增等繁琐的操作步骤,同时也避免了预扩增过程中所带来的偏向性。
基于SHERRY方法的转录组测序建库流程如图1所示,样本可以来自裂解的单细胞或bulk RNA;RNA分子经过oligo-dT 引物的反转录后,RNA/DNA杂合链可直接被Tn5转座酶打断(图中灰色的波浪线及直线分别代表RNA及DNA分子);在Tagmentation之后,转座酶会在杂合链两端加上adaptor接头,进行之后的建库PCR扩增。
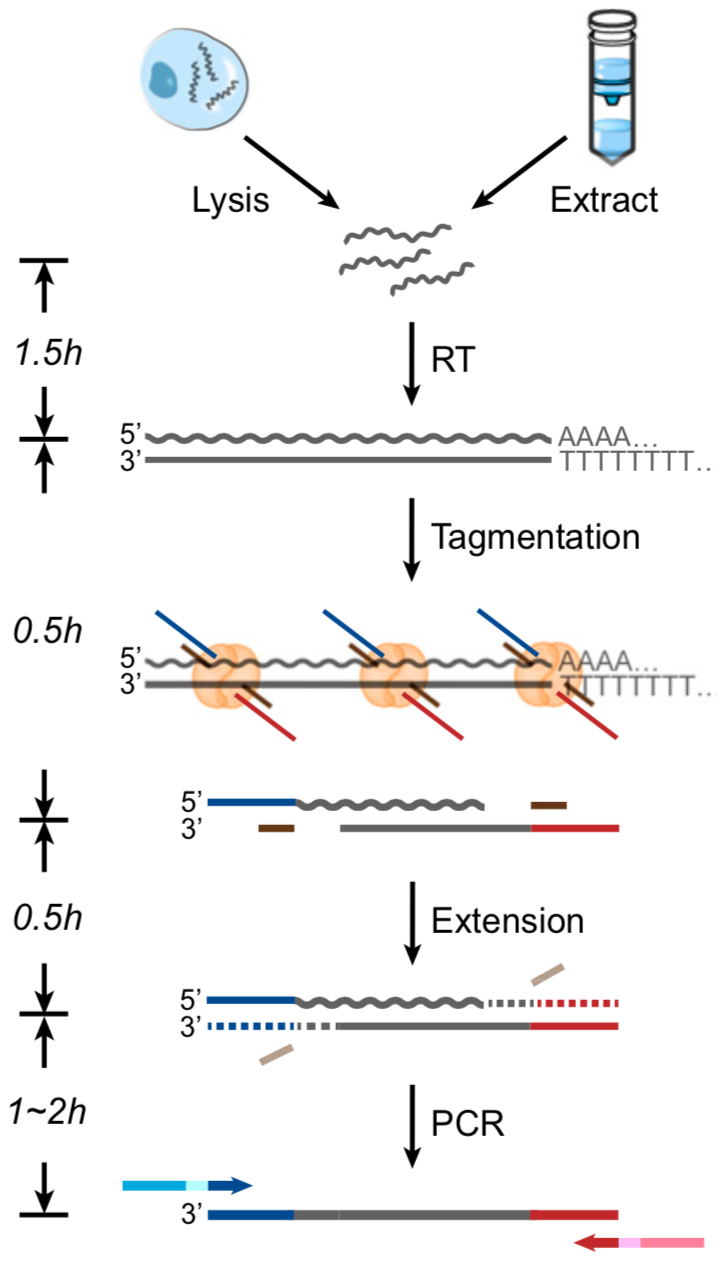
图1 SHERRY转录组测序建库流程
基于100pg的RNA起始量,SHERRY表现出了高的read匹配率,同时可检测出近9000个基因,其中72%的基因可在三个重复样本中检测出,可重复性较好。与目前普遍应用于单细胞转录组测序的Smart-seq2技术相比,SHERRY与其建库质量不相上下,并且具较低的GC bias。此外,SHERRY整个流程仅需要5个步骤,且可在一个管子中完成,整个时间仅约4小时,其中手工操作时间不到半小时。相比之下,Smart-seq2整个耗时约为SHERRY二倍的时间,且需要一些前处理等操作;此外,包含十个步骤的NEBNext方法则需要更加多的实验室操作和时间成本。值得一提的是,在成本方面,SHERRY方法仅为其他两种方法的五分之一,将大大节省实验成本。
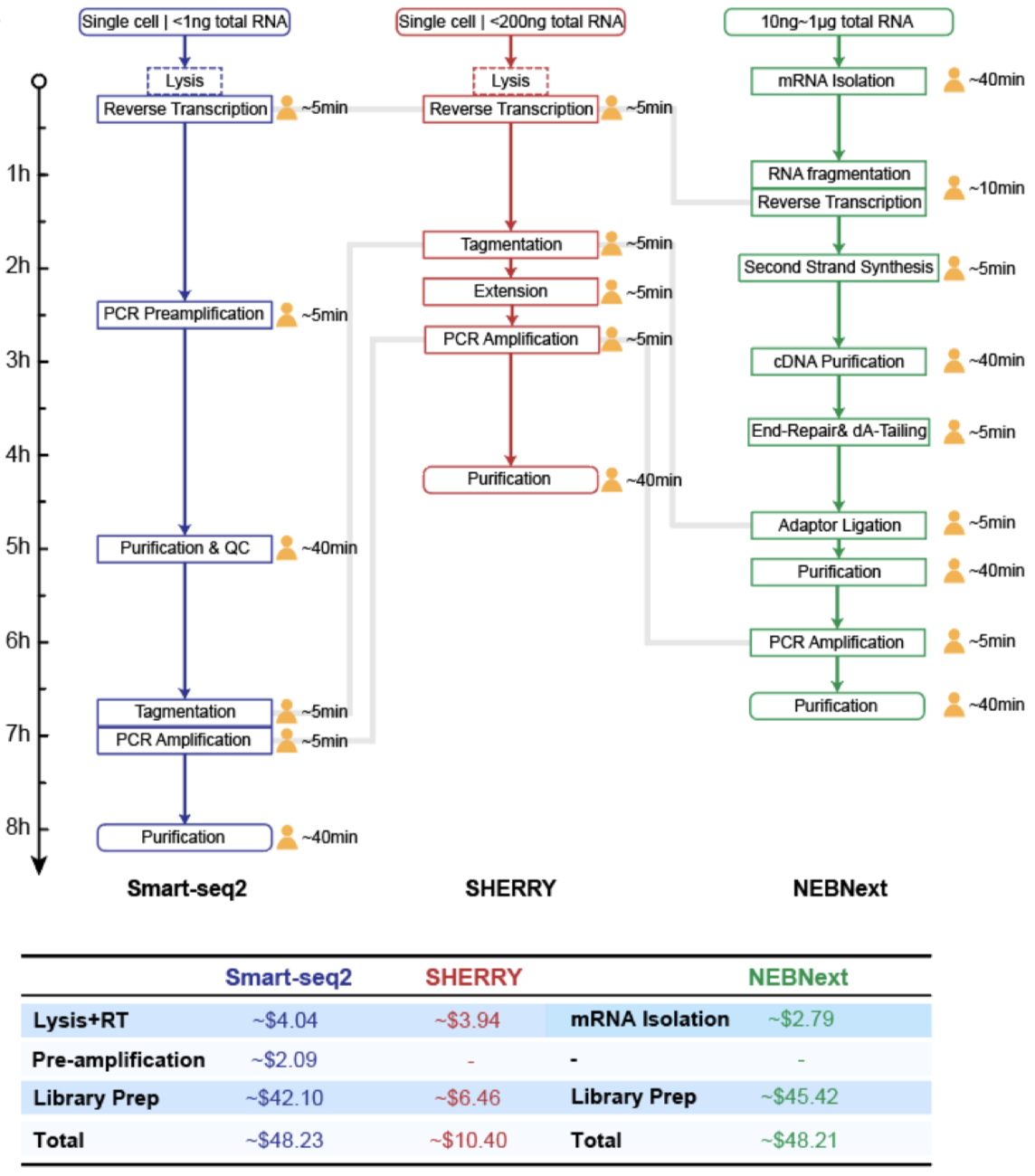
图2:Smart-seq2, SHERRY 及NEBNext流程及成本比较
文章通讯作者之一,北京大学谢晓亮教授指出:“虽然SHERRY 应用广泛,当下我们第一个临床应用是对nCoV感染者进行测序。nCoV病毒属于RNA 病毒,其序列已被中国科学家测出,从而有了刚刚获得药监局批准的几家nCoV检测试剂盒。这些试剂盒利用 RT PCR 的方法对病毒的某一位置定点测量。而这两万九千碱基的病毒突变很多,且随时间变化,因而亟需测序。SHERRY应该比现有的RNA 测序建库方法更灵敏,更简单。这对nCoV的检测和分析十分重要,因为我们希望避免在高封密(P3 和 P4)实验室中的RNA 富集操作。我们的团队正在夜以继日,争分夺秒地攻关,希望提供更好的nCoV 测序方法。”
该论文的两位共同第一作者是北京大学生物医学前沿创新中心(BIOPIC)的博士研究生狄琳和傅语思,大部分的实验工作在北京大学和清华大学合作下完成。这一工作的合作者还包括南京师范大学王冠博教授、美国XGen公司劳开勤博士团队以及美国加州大学圣地亚哥分校王栋教授团队。该工作得到了国家自然科学基金、国家科技部、北京未来基因诊断高精尖创新中心(ICG)、北京结构生物学高精尖创新中心、北大-清华生命科学联合中心、北京脑科学计划以及深圳湾实验室的支持。
原文链接:RNA sequencing by direct tagmentation of RNA/DNA hybrids